DNA- Analyse
1. Einführung
2. Biologische Grundlagen der DNA-Analyse
3. Technik der DNA-Analyse
4. DNA-Analyse mit dem Blue Genes Koffer
5. Auswertung und Diskussion
6. Literatur
7. Bezugsquellen
1. Einführung
Jedem Krimi- oder interessierten Zeitungsleser ist heute bekannt, dass ein Straftäter fast immer biologische Spuren hinterlässt. Im Normalfall ist es dann der "Strichcode" seiner Erbsubstanz, verpackt in einer einzigen Zelle, die der Spurenleger hinterlassen hat. Ein Waffenbesitzer kann schlecht leugnen, die Tatwaffe in der Hand gehabt zu haben, nachdem wenige Zellen seines Hautabriebes am Griff der Waffe gefunden wurden [ 1] . Sogar ein Einbrecher, der zwar sorfältig auf das Nichtvorhandensein von Fingerabdrücken achtet, verliert garantiert auch einmal ein Haar. Das reicht, denn sein "unveränderliches Kennzeichen" befindet sich darin. Um auch noch die biologische Spur zu bestätigen, kann er am Tatort eine Zigarette rauchen. An der hinterlassenen Zigarettenkippe befinden sich dann genug Mundschleimhautzellen, die sein "Kennzeichen" verdeutlichen. Eine Speichelprobe des Verdächtigen als Vergleichssubstanz bestätigt das "Kennzeichen".
Auch ein ganz raffinierter Doping-Sünder, der zu seiner Entlastung den falschen Urin abgibt, wird in Schwierigkeiten geraten, wenn in der Flüssigkeit die falschen Zellen zu finden sind.
Es ist auch bekannt, dass die DNA-Analyse für eindeutige Verwandtschaftsbeziehungen von großem Wert ist. In Zweifelsfällen konnte bis vor wenigen Jahren nur aufgrund dringender biologischer Indizien einem Mann eine Vaterschaft nahegelegt werden. Heute bringt eine DNA-Typisierung eine Sicherheit von eins zu vielen Millionen, ob eine Vaterschaft vorliegt oder nicht.
Auch konnten viele interessante Verwandtschaftsfragen gelöst werden: Die DNA von Knochen aus dem Grab der russischen Zarenfamilie zeigte, dass die einzige noch lebende angebliche Tochter Anastasia tatsächlich nicht aus der Herrschaftsfamilie stammt. Auch ist nun endgültig klar, dass das Findelkind Kaspar Hauser nicht wegen eines Streites um die Erbfolge einer Adelsfamilie im Wald ausgesetzt wurde. Alte Blutflecken an der Kleidung Kaspar Hausers, welche in einem Heimatmuseum aufbewahrt wurde, beweisen, dass die DNA nicht aus nämlichen blauen Geblüt stammt.
Die DNA-Typisierung wird nicht nur beim Menschen durchgeführt. Bei einem japanischen Einzelhändler wurde Fleisch untersucht . Es wurde herausgefunden, dass das Fleisch von Buckelwalen stammt, einer Tierart, die seit 1966 streng geschützt ist. Bemerkung am Rande: Um der Strafe zu entgehen, behauptete der Händler, das verbotene Fleisch sei vor der Zubereitung 30 Jahre eingefroren gewesen [2].
2. Biologische Grundlagen der DNA-Analyse
Die komplette Bauanleitung für unseren ganzen Körper ist in jedem einzelnen Zellkern der Zellen, aus denen wir bestehen, gespeichert. Also in jeder Haut-, Speichel- oder Spermienzelle. Diese Information ist verschlüsselt in einem Code, d.h. einer Art chemischer Buchstabenfolge (Sequenz) von nur vier verschiedenen Molekülen. Das sind die Basen mit den Namen Adenin, Guanin, Thymin und Cytosin. Die Besonderheit ist nun die, dass nur vier Prozent der rund drei Milliarden Basen codierend sind. Das heißt, nur hier befinden sich die eigentlichen Gene bzw. die wichtigen Erbinformationen. Die restlichen 96 % bestehen aus nicht codierender DNA. Das bedeutet, dass dieser größere Abschnitt keinen Bauplan für unseren Körper enthält. Einerseits verrät dieser Abschnitt also nichts über Psyche, Intelligenz oder Krankheit einer Person. Andererseits ist dieser Abschnitt in bestimmten Bereichen von Mensch zu Mensch verschieden ohne Auswirkung nach außen. Genau diese Individualisierung von DNA-Bereichen ist für die DNA-Analyse geeignet.
Man findet dort Bereiche, in denen sich kleine DNA-Grundeinheiten von wenigen Basenpaaren verschieden oft wiederholen. Man bezeichnet einen solchen Genomabschnitt (Locus) mit dieser Eigenschaft als "Variable Number of Tandem Repeats" (VNTR)-Region.
Vereinfachte Darstellung eines VNTR-Locus mit 7 Wiederholungen:
| Flankierende DNA-Sequenz 1 | TCTT | TCTT | TCTT | TCTT | TCTT | TCTT | TCTT | Flankierende DNA-Sequenz 2 | ||
| (Bei allen Menschen gleich) |
Wiederholung kleiner DNA-Grundeinheiten |
(Bei allen Menschen gleich) | ||||||||
Ein solcher Abschnitt sei z.B. das Allel 22 des Systems FGA. Dieses System heißt so, weil es sich am a -Fibrinogen-Gen befindet. Das Allel 22 enthält 22 der typischen Wiederholungen von je 4 Basenpaaren. Mit den dazu gehörenden , einrahmenden Sequenzen kommt dieser DNA-Abschnitt dann auf eine Größe von 196 Basenpaaren. Dieser DNA-Abschnitt kann durch PCR und Elektrophorese nachgewiesen werden indem an einer bestimmten Stelle ein kleiner Strich erscheint. Das Allel 24 des Systems FGA besitzt zwei Wiederholungen mit den vier Basenpaaren mehr und ist folglich 204 Basenpaare groß und ein weiteres PCR-Signal erscheint in der Elektrophorese.
Jede kernhaltige Körperzelle hat nun mal zwei Allele des Systems FGA. Das eine Allel stammt ursprünglich von der Mutter; das andere vom Vater. Wurde bei dem System FGA eine Typisierung "22/24" bei der DNA-Analyse ermittelt, so bedeutet dies, dass ein Elternteil das Allel 22, und das andere das Allel 24 beisteuerte.
Neben dem System FGA werden auch noch andere Systeme mit ihren charakteristischen VNTR's verwendet. Das sind z.B. FES, THO1, vWA, SE33 oder D8S306. Ihre Namen beschreiben den Ort, wo sie im Genom gefunden wurden. D8S306 liegt auf Chromosom 8 am "Kilometerstein" 306; vWA liegt neben dem Von-Willebrand-Faktor. Liegt ein System auf einem Geschlechtschromosom, so zeigt die DNA-Analyse ob das DNA-Material von einer Frau oder einem Mann stammt. Es liegt auf der Hand, dass je mehr Systeme man zur DNA-Analyse einsetzt, desto aussagekräftiger die Analyse ist.
Eine Gelelektrophorese für einen Vaterschaftsnachweis nur mit dem System FGA könnte z.B. so aussehen:
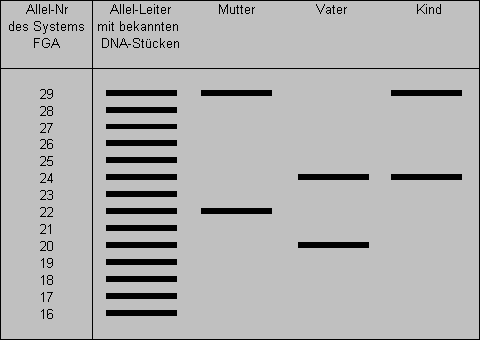
DNA-Typisierung .........................22/29 ...............20/24 ................24/29
Das Allel 24 des Kindes könnte vom Vater stammen. Selbstverständlich müssen bei einem zuverlässigen Vaterschaftsnachweis weitere Systeme herangezogen werden. Auch muss die Wahrscheinlichkeit mit einbezogen werden, wie oft das Allel in einer bestimmten Bevölkerungsgruppe auftaucht.
Angenommen, ein Spurenleger hinterlässt auf einer Tatwaffe einige wenige Hautzellen. In den Zellen wurde folgende DNA-Typisierung mit den 5 Systemen FES, THO1, vWA, FGA und SE 33 gefunden. Seine Speichelprobe ergibt die selbe Typisierung. Wie groß ist nun die Wahrscheinlichkeit, dass nicht vielleicht ein anderer Mensch die gleiche Typisierung aufweist?
|
System |
Typisierung (Allelbestimmung) z.B. |
Häufigkeit in einer Bevölkerungs- gruppe in Prozent |
Häufigkeit der Allelkombination (a x b x 2) für Heterozygotie a2 für Homozygotie |
|
FES THO1 vWA FGA SE33 |
10/11 6/9 14/19 20/25 15/22 |
29,8 / 43,4 24,5 / 15,2 9,3 / 9,2 13,4 / 7,9 4 / 0,9 |
25,7 7,4 >1,7 2,1 0,07 |
Werden nun die Multiplikationsergebnisse der 5 kombinierten Systeme miteinander multipliziert, so erhält man einen statistischen Häufigkeitswert, der angibt, wie oft dieses DNA-Muster innerhalb der nicht verwandten Bevölkerung zu erwarten ist. In unserem Ergebnis: 5 x 10-9 , was bedeutet, dass unter zirka einer Milliarden Menschen statistisch betrachtet nur einer dieses "Kennzeichen" haben kann.
3. Technik der DNA-Analyse
3.1. Probengewinnung
Zunächst werden die zu untersuchenden Zellen in ein PCR-Reaktionsgefäß gebracht. Darin wird anschließend die Erbsubstanz in Lösung herausgelöst.
3.2. PCR
Wie werden die Allele eines VNTR-Systems sichtbar gemacht? Dazu bedient man sich der Polymerasekettenreaktion (PCR), für deren Entwicklung Kary B. Mullis im Jahre 1993 den Nobelpreis erhielt. Mit der PCR können winzigste Mengen eines DNA-Abschnitts so stark vervielfältigt werden, dass man sie nach Anfärben sehen kann.
Dazu braucht man zunächst einmal Primer. Primer sind selbst kurze DNA-Abschnitte, die als Gegenstück genau auf die die VNTR-Loci flankierenden DNA-Bereiche passen. Diese Anlagerung findet bei einer bestimmten Temperatur sehr schnell statt. Von den Primern aus findet dann die Synthese des gewünschten DNA-Abschnittes durch das Enzym Taq-Polymerase statt. Eine Vervielfältigung des DNA-Abschnittes erfolgt in Zyklen. Bei jedem Zyklus erfolgt eine Verdoppelung des DNA-Abschnittes. Nach 30 Zyklen liegen dann theoretisch 230 Kopien vor.
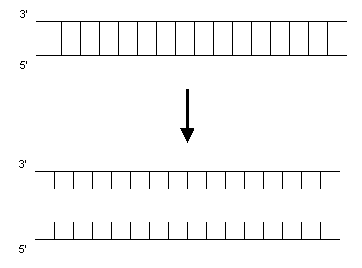
Ein Zyklus besteht aus drei Teilschritten:
1. Zuerst wird der DNA-Doppelstrang bei 95 °C denaturiert:
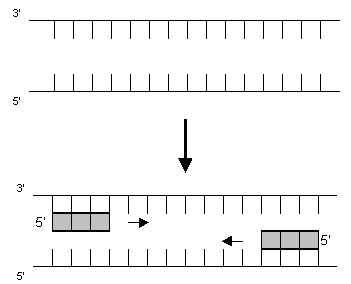
2. Anschließend werden die beiden Primer bei 60 °C angelagert (Hybridisierung):
3. Das Enzym Taq-Polymerase ergänzt nun bei 72 °C die DNA-Einzelstränge indem sie die DNA-Bausteine komplementär entsprechend der vorhandenen Matrize zusammenfügt (Basenpaarregel):
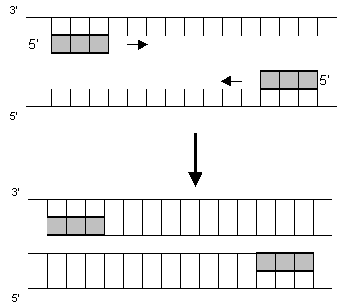
Schon nach wenigen Zyklen wird nur noch der Bereich vervielfältigt, der von den Primern eingerahmt wird. Natürlich werden die Primer so konstruiert, dass sie den gewünschten VNTR-Locus einrahmen.
Die Zyklen laufen automatisch in einem Thermocycler ab. Ein Thermocycler ist ein Heizgerät, das nach entsprechender Programmierung das Reaktionsgemisch in bestimmten Zeiten auf eine gewünschte Temperatur bringt. Das Reaktionsgemisch enthält alle benötigten Komponenten: die zu untersuchende DNA (Template), die für den VNTR-Locus ( die Systeme FGA, THO1 oder andere) verantwortlichen beiden Primer, die DNA-Bausteine (Nukleotide), den Reaktionspuffer und das Enzym Taq-Polymerase.
3.3. Elektrophorese
Ist durch die PCR genügend DNA-Material eines Allels (VNTR-Locus) hergestellt worden, so kann mittels Gelelektrophorese die DNA-Länge (Repeat-Anzahl) bestimmt werden. Dabei werden die unterschiedlich großen DNA-Stücke längenabhängig aufgetrennt und anschließend durch Färbung sichtbar gemacht
Zunächst wird ein Polyamidacryl-Gel gegossen, welches man sich als ein System von feinen Poren vorstellen kann. Das PCR-Produkt wird in kleine Geltaschen gefüllt. Durch die feinen Poren wandern dann die DNA-Stücke im elektrischen Feld. Sie tun dies, weil sie in einem Puffer negativ geladen sind.
Je kleiner die Fragmente sind, desto schneller wandern sie durch die feinen Poren.
Wie weit die Wanderung im Gel fortgeschritten ist, erkennt man an einem Farbstoff (Bromphenolblau und Xylencyanol), den man mitwandern lässt. Die DNA-Stücke werden zum Schluss durch Anfärben mit Silber sichtbar gemacht.
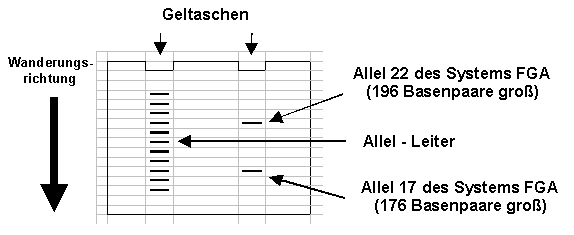
Um die Größe der Allele zuordnen zu können, lässt man ein Gemisch aus DNA-Fragmenten mitwandern, deren Größe bekannt ist. Dieses Allelcocktail wird auch Allel-Leiter genannt.
4. DNA-Analyse mit dem Blue Genes Koffer (1)
4.1. Vorbemerkung
Die in der DNA-Analyse verwendeten Systeme wie vWA, THO 1, FGA oder FES liefern DNA-Fragmente in der Größenordnung von 100 bis 240 bp. Die Allele unterscheiden sich oft nur in 4 bis 6 bp. Um eine verwertbare Analyse zu erhalten, muss mit einem hochauflösenden Gel elektrophoretisch getrennt werden. Da Polyamidgele engmaschiger sind als Agarosegele, verwendet man diese in der Praxis. Die Banden erscheinen schwarz weil die DNA mit Silber gefärbt wurde.
Das Trenngel des Blue Genes Koffer ist allerdings Agarose. Außerdem wird als Färbemittel AzurB-Chlorid anstatt dem empfindlicheren, aber giftigen Ethidiumbromid verwendet. Um ein Ergebnis zu erhalten, wurde im beschriebenen Experiment auf das System D1S80 ausgewichen, dessen DNA-Fragmente 400 – 800 bp groß sind (3,4). Man erreicht so eine individuelle Typisierung. Allerdings können nicht immer 2 Allele erkannt werden, da die Wiederholungssequenzen (Short tandem repeats, STR) nur 16 bp groß sind.
4.2. Materialen
Zusätzlich zum Blue Genes Koffer werden benötigt:
- Thermocycler (6)
- Ready-Mix (10,11)
- Paraffinöl (8)
- PCR-Reaktionsgefäße (7)
- Sterilhandschuhe
- Primer für D1S80 (5)
- InstaGene Matrix (15)
- Steriles Wasser
- Physiologische Kochsalzlösung
- Dampfdrucktopf (Sterilisation)
- Vortex-Gerät (Gerät zum Durchmischen der Proben) (14)
- Zentrifuge (14)
- Wattestäbchen und Skalpell (alternativ zur physiologischen Kochsalzlösung)
- eventuell: BSA ( bovine serum albumin) (15)
- Pipette für 200 µl (9)
4.3. Versuchsdurchführung
4.3.1. Probengewinnung
1. Mit Wattestäbchen
- Mit einem sterilen Wattestäbchen wird ein Abstrich von der Mundschleimhaut gemacht. Am besten streicht man damit mehrmals zwischen Lippe und Zahnfleisch oder an der Wange entlang. Man lässt das Wattestäbchen über Nacht trocknen.
- Vom eingetrockneten Wattestäbchen wird mit einem sterilen Skalpell ein ca. 5 mm großes Stück abgeschnitten und in ein Eppendorf-Gefäß gebracht.
2. Mit physiologischer Kochsalzlösung
- Mund mit Wasser kurz ausspülen
- 10 ml physiologische Kochsalzlösung 10 s lang gurgeln
- 1 ml davon in ein Eppendorf-Gefäß geben und 10 s lang bei maximaler Geschwindigkeit zentrifugieren
- Überstand verwerfen und den Niederschlag in 1 ml physiologischer Kochsalzlösung wieder aufnehmen. Es wird mit dem Vortexgerät resuspendiert und erneut zentrifugiert.
- Überstand verwerfen. Der Niederschlag (Pellet) enthält die gewünschten Mundschleimhautzellen.
3. Vorbereitung der Proben für die PCR
InstaGene Matrix enthält eine Suspension des Ionenaustauschers Chelex, welcher Magnesiumionen bindet. Magnesiumionen beeinflussen zum einen die PCR, und zum andern katalysieren sie enzymatische Reaktionen zum Abbau von DNA.
- Die InstaGene Matrix wird durchmischt (Vortex-Gerät). Davon werden 200 µl zur Watte mit Speichel aus a) oder zum Pellet aus b) gegeben. Anschließend wird mit dem Vortexer geschüttelt.
- Um DNase zu inaktivieren wird die Probe 30 min auf 56 °C erhitzt (Wasserbad oder Thermocycler). Danach wird mit dem Vortex-Gerät geschüttelt.
- Die Membranen der Mundschleimhautzellen werden danach durch Erhitzen auf 100 °C ( 8 min ) zerstört. (Deckel gegen Aufspringen sichern!)
- Anschließend wird nochmals mit dem Vortexer geschüttelt und danach 1 min bei maximaler Geschwindigkeit ( 15000 rpm ) zentrifugiert. Der Überstand enthält die gewünschte DNA für die PCR. (Vorsicht, Chelex darf nicht in den PCR-Ansatz gelangen !)
4.3.2. PCR
Die Sequenzen [ 4] der Primer für D1S80 heißen ( 5‘ – 3‘ ):
OF: GAA ACT GGC CTC CAA ACA CTG CCC GCC G
OR: GTC TTG TTG GAG ATG CAC GTG CCC CTT GC
Um ein besseres PCR-Ergebnis zu erzielen, wird oft noch BSA zugestzt.
PCR – Ansatz:
|
Komponente |
Konzentration |
Volumen |
|
Ready-Mix (enthält: Taq-Polymerase, Mg2+, Puffer) Primer OF Primer OR BSA |
2x 25 pmol/µl 25 pmol/µl 0,1 % |
25 µl 2 µl 2 µl 10 µl |
|
Aqua dest. DNA – Probe |
9 µl 2 µl |
|
|
Paraffin-Öl |
1 Tropfen |
Temperatur – Zeit – Programm für die PCR:
|
Temperatur |
Zeit |
Reaktionsschritt |
Zyklen |
|
94 °C |
3 min |
Denaturieren |
|
|
94 °C |
1 min 30 s |
Denaturieren |
|
|
65 °C |
1 min 30 s |
Hybridisieren |
40 x |
|
72 °C |
1 min 30 s |
Polymerisieren |
|
|
72 °C |
10 min |
Polymerisieren |
|
|
4 °C (wenn möglich 4 °C, bzw. einfrieren) |
bis zur Gelelektrophorese |
4.3.3. Elektrophorese
Entsprechend der Blue Genes Anleitung wird die Gelelektrophorese durchgeführt. Jedoch wird ein 2 %-iges Gel hergestellt und bei 95 V ca. 20 min getrennt.
5. Auswertung und Diskussion

Bild 1: Zuordnung des PCR-Produkts zu Marker III
Zur Bestimmung der Fragmentgröße des PCR-Produkts wurde noch eine Elektrophorese mit 1 %-igem Agarosegel ( 95 V, 15 min ) durchgeführt. Vergleicht man die Banden von Bild 1 mit dem entsprechenden Bild aus der Blue Genes Anleitung ( S. 91), so kann man auf eine Fragmentgröße von 500 bis 800 bp schließen.
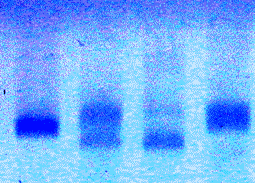
Bild 2: DNA-Analyse mit D1S80
Die Spuren 1 bis 4 in Bild 2 stammen von vier verschieden Personen. Durch die höhere Auflösung mit 2 %-igem Agarosegel ist eine individuelle Typisierung klar zu erkennen. Bei der Person zu Spur 2 ist eine Alleltrennung deutlich. Da es sich hier um das Kind der Personen 3 und 4 handelt, lassen sich sogar die vererbten Allele zuordnen.
6. Literatur
[ 1] W.PFLUG, T.M.H. NGUYEN, J. MERKEL: Zuordnung von Schusswaffen mittels DNA-Analyse.- Kriminalistik 12/97 – Hüthig Fachverlag Heidelberg
[ 2] M. BENECKE: Kriminalbiologie – BLT/Lübbe Bergisch Gladbach 1999
[ 3 J.SCHEE-GRIESE, G.BLÄß, S. HERRMANN, H.R. SCHEIDER, R. FÖRSTER, G. BÄßLER and W. PFLUG: Frequence distribution of D1S80 alleles in the German Pupulation – Forensic Science International, 59 (1993) 131 – 136 - Elsevier Scientific Publishers Ireland Ltd.
[ 4] Daniel B. Demers, Elizabeth T. Curry, and Amanda C. Sozer : Improved PCR Amplification of VNTR Locus D1S80 Using PNA(Peptide Nucleic Acid) Oligomers - http://www.promega.com/geneticidproc/ussymp6proc/demers.htm
7. Bezugsquellen
(1) Blue Genes Kit: Fonds der Chemischen Industrie Karlstr. 21, 60329 Frankfurt
(3) Komplettes Reagenzienset für den Genbaukasten, bestellbar beiRoche Diagnostic Boehringer Mannheim GmbH, Molekular Biochemicals D-68298 Mannheim Tel 0800/759 2226
(4) DNA-Molekulargewichtsmarker III, Artikelnr. 528552, einzeln bestellbar als Reagenz des Genbaukastens unter dem Stichwort "Blue Genes" bei Roche Diagnostic Boehringer Mannheim GmbH, Molekular Biochemicals D-68298 Mannheiom Tel 0800/759 2226
(5) Die beiden Primer kann man z.B. bei der Fa. amersham pharmacia biotech GmbH, Postfach 5480, D-79021 Freiburg synthetisieren lassen. oligo.ordering@eu.apbiotech.com.Auch können die Primer bei der Fa. SIGMA ARK (Fax: 0695 007 0306; sigma-ark@eurnotes.sial.com synthetisiert werden.
(6) Es gibt sehr viele Anbieter: z.B. BIO-RAD laboratories GmbH, Postfach 450133, 80901 München; Biometra GmbH, Rudolf Wissell Str. 30, D-37079 Göttingen;Eppendorf-Netheler-Hinz GmbH, Friedensstr.116, 51145 Köln
(7) z.B. Multiply-Pro, 500 m l, Best.Nr. 72.735, 500 Stck./Beutel,Preis ca. 60.- . Bestellbar bei Sarstedt AG & Co. , Postfach 1220, D-51582 Nümbrecht
( 8) Paraffinöl (mineral oil) Best.Nr. 69794, 100 ml, Preis ca. 20.-, Fluka Sigma-Aldrich Chemie GmbH Grünwaldweg 3, 82041 Deisenhofen
(9) Eppendorf-Netheler-Hinz GmbH, Friedensstr.116, 51145 Köln
(10) Promega GmbH, High Tech Park, Schildkrötstr. .15, 68199 Mannheim
(11) Sigma-Aldrich GmbH, Eschenstr. 5, 82024 Taufkirchen
(12) Amersham Pharmacia Biotech GmbH, Postfach 5480, 79021 Freiburg
(13) Sigma Ark (Fax: 0695 007 0306; sigma-ark@eurnotes.sial.com )
(14) Carl Roth GmbH, Postfach 100121, 76231 Karlsruhe
(15) BIO-RAD laboratories GmbH, Postfach 450133, 80901 München
Autor:
StD Rudolf Heinze, Keltenstr. 74/2, 73773 Aichwald
bearbeitet von Ursula Gegler-Hertler



