PCR als Schulexperiment
1. Einleitung
2. Grundlagen
3. Zeitschema
4. Materialien
5. Versuchsdurchführung
6. Ergebnis und Diskussion
7. Bezugsquellen
8. Literatur
9. Kostenübersicht
10. Zeit-Protokoll
11. Links zu Anwendungsbeispielen von 
Vorbemerkung
Die Polymerasekettenreaktion (PCR)* entwickelte sich zu einer der wichtigsten Methoden in der Molekularbiologie. Diese und weitere molekularbiologische Arbeitsmethoden werden in verschiedenen Lehrbüchern der Biologie beschrieben [1] . Es soll ein PCR-Experiment gezeigt werden, das mit den an Schulen häufig vorhanden Experimentierkits, wie dem Lamda-Kit oder dem Blue Genes Kit durchgeführt werden kann. (1)(2)
*Die PCR ist patentrechtlich geschützt für Hoffmann-La Roche
Autor: StD Rudolf Heinze, Keltenstr.74/2 73773 Aichwald
Webbearbeitung: Ursula Gegler
1. Einleitung
Im Jahre 1992 erhielt Kary B. Mullis den Nobelpreis für die Erfindung der Polymerasekettenreaktion. Mit Hilfe der hitzestabilen Taq-Polymerase, welche aus in heißen Quellen lebenden Bakterien (Thermus aquaticus) isoliert wurde, wurde es möglich, einen bestimmenten Abschnitt einer DNA in kurzer Zeit millionenfach zu vermehren. Der
vervielfältigte Abschnitt kann dann durch Gelelktrophorese sichtbar gemacht werden oder für weitere Experimente, wie z.B. Klonierungsversuche weiter verwendet werden. Welcher Abschnitt vermehrt wird, bestimmt der Experimentator, indem er den gewünschten Bereich durch "Primer" eingrenzt. Primer sind kurze DNA-Stücke, die komplementär auf die Ziel-DNA passen.
Die PCR revolutionierte innerhalb kurzer Zeit die Molekularbiologie. Ungeahnte Möglichkeiten in der Verwendung öffneten sich. In der medizinischen Diagnostik können mit der PCR schnell und sicher Infektionen durch Viren, Bakterien, Pilzen oder Einzeller nachgewiesen werden sofern Bereiche ihrer Erbsubstanz bekannt sind. Da die PCR höchst sensitiv und schnell ist, kann z.B. das AIDS-Virus nachgewiesen werden lange bevor ein immunologischer Nachweis erfolgen kann . Auch in der Grundlagenforschung spielt sie eine dominierende Rolle, sei es bei der Sequenzanalyse, der gezielten Mutagenese oder sei es bei der zellunabhängigen Klonierung [2] [3] [4] . Weit mehr bekannt wurde die PCR durch die DNA-Analyse [5] . Durch gezieltes Vermehren von DNA-Abschnitten, die von Mensch zu Mensch verschieden lang sind, erhält man bei der anschießenden Gelelektrophorese bestimmte Muster vergleichbar einem Strichcode. In der Kriminalbiologie können so Täter, die irgend welche Zellen als Spuren hinterlassen haben, leicht identifiziert werden. Nach dem gleichen Prinzip werden Verwandtschaftsbeziehungen geklärt. Werden Bestandteile der Nahrung oder Kleidung auf die biologische Herkunft hin untersucht, bedient man sich auch der PCR.
Im folgenden Experiment wird mittels PCR die DNA des Bakteriophagen lambda nachgewiesen. Dazu wird ein 799 bp großes DNA-Fragment aus dem,Bakteriophagen vervielfältigt, durch Gelelektrophorese getrennt und dann durch Anfärben sichtbar gemacht. Lambda-Phagen-DNA eignet sich für Schülerversuche besonders gut, da diese DNA preisgünstig zu kaufen ist .
2. Grundlagen
Prinzip der PCR
Die Vervielfältigung eines Abschnitts aus einem DNA-Doppelstrang erfolgt in zyklischen Wiederholungen [7] [8] . Die spezifischen Primer (einzelsträngige Oligonukleotide aus 15 bis 30 Nukleotiden) bestimmen den zu vervielfältigenden Abschnitt. Bei jedem Zyklus erfolgt eine Verdoppelung des DNA-Abschnitts, d.h. nach 30 Kopien liegen 230 Kopien vor.
Die drei Teilschritte eines Zyklus sind:
a) Denaturieren: Trennen des DNA-Doppelstrangs bei 95 °C .
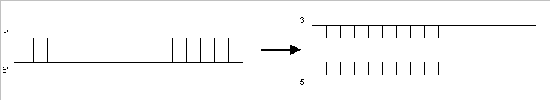
b) Hybridisieren: Anlagern (Annealing) der beiden Primer* an die DNA-Einzelstränge bei 58 °C. (Die Temperatur wird durch den Gehalt an G-und C-Nukleotiden der Primer bestimmt [7])
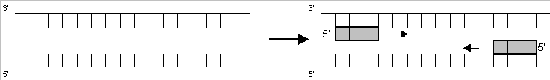
c) Polymerisieren (Extension): Ergänzung zu einem DNA-Doppelstrang durch die Taq-Polymerase bei 72 °C.
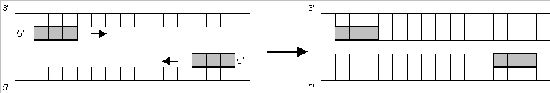
Im nächsten Zyklus werden die beiden DNA-Stränge wieder getrennt, die Primer angelagert und mittels Taq-Polymerase wieder zu DNA-Doppelsträngen ergänzt.
Die in genügender Menge vorliegenden DNA-Abschnitte können durch Elektrophorese getrennt und durch Anfärben sichtbar gemacht werden.
3. Zeitschema für den Versuchsablauf:
| Zeitbedarf |
Anschließende Lagerung möglich bei minus 18°C |
Zeit für Theorie |
||
| Vorbereitung der Reagenzien | Vorbereitung des Arbeitsplatzes |
15 min |
||
| Ansetzen der Stammlösungen |
30 min |
x |
||
| PCR | Vorbereitung Wasserbäder oder Thermocycler
Auftauen der PCR-Komponenten |
15 min |
||
| Pipettieren der PCR-Ansätze |
15 min |
|||
| PCR - Cycling mit Wasserbädern |
50 min |
x |
||
| (PCR - Cycling mit Thermocycler) |
90 min |
x |
(90 min) |
|
| Elektrophorese | Vorbereitung des Gels und der Proben |
60 min |
||
| Auftragen der Proben auf das Gel |
15 min |
|||
| Elektrophorese mit Lambda-Kit, 25 - 30V |
90 min |
60 min |
||
| (Elektrophorese mit Blue Genes Koffer, 90V) |
(30 min) |
|||
| Färben und Entfärben des Gels |
40 min |
30 min |
||
Tabelle 1: Zeitbedarf für den Versuch
4. Materialien
Aus dem "Blue Genes" Koffer oder dem Lambda-Kit werden verwendet (1)(2):
- Elektrophoreseapparatur (Netzgerät und Kammer)
- Eppendorf-Mikropipette (Blue Genes Koffer, 2-20 µl) bzw. Mikropipette (Lambda-Kit, 2,10 l)
- DNA-Auftragspuffer
- TBE-Elektrophoresepuffer oder TAE-Puffer
- Agarose
Aus der zum "Blue Genes" Kit gehörenden Reagenzienbox stammen (3):
- steriles Wasser (kann aber auch selbst hergestellt werden), AzurB-Chlorid (DNA-Färbemittel oder Färbemittel aus Lambda-Kit ; bzw. Sicherheitsfärbemittel von BIO-RAD (12)
- DNA-Marker III (es kann aus Kostengründen darauf verzichtet oder ein anderer Längenstandard verwendet werden)(4)
Zusätzlich wird benötigt:
- 70%-iger Spiritus (beim Färben mit Lambda-Kit)
- 1 Mikropipette, 2-20 µl ( falls nur Lambda-Kit vorhanden)
- Primer OF und OR (5)
- Lambda-DNA (14)
- Ready-Mix (enthält Taq-Polymerase, Puffer, MgCl2, Nukleotide) (13)(14)
- Paraffinöl (mineral oil) (9)
- Laborübliche Glasgeräte
- PCR-Reaktionsgefäße ( 0,5 ml ) (8)
- 3 Wasserbäder (95 °C, 62 °C, 72 °C). Dazu eine Pinzette und eine schwimmfähige Halterung aus einer 6 mm starken StyroporR-Tapetenisolierung (5 x 3 cm), in die mit einem Korkbohrer entsprechende Löcher gestanzt wurden. Alternativ: Thermocycler (7)
- Stoppuhr
- Sterilhandschuhe
- Dampfdrucktopf zum Sterilisieren von Pipettenspitzen und Reaktionsgefäßen (in Bechergläsern, mit Abdeckung aus Alu-Folie )
- Petrischale oder Styroporbox mit Eis
- Netzgerät oder Batterien (wenn nur Lambda-Kit)
- Ständer für Reaktionsgefäße (z.B. selbst gebastelt: Plexiglas- oder Hartschaumblock mit Bohrungen für die PCR-Reaktionsgefäße).
5. Versuchsdurchführung
5.1. Reaktionsansatz für die PCR
Hinweis: Steril arbeiten! Biochemikalien bei – 20 °C aufbewahren ! (Ready-Mix im Kühlschrank)
Entsprechend Tabelle 2 werden die Reaktionsansätze in die speziellen PCR-Gefäße pipettiert.
Nach Zusammenmischen aller Komponenten muss sofort mit dem Cycling (Wasserbäder oder Thermocycler) begonnen werden.
Die Stammlösungen sind berechnet für 10 Reaktionsansätze ( incl. Negativkontrolle, für 5 Arbeitsgruppen) und können nach Bedarf variiert werden. Auch wurde ein "Pipettierschwund" berücksichtigt.
Bis auf die Taq-Polymerase können die Lösungen rechtzeitig vorbereitet und bei – 20 °C längere Zeit aufbewahrt werden.
Auch wurde berücksichtigt, dass mit den einfachen Pipetten des Lambda-Kits nur 2 bzw. 10 µl abgemessen werden können.
| Komponente | Lieferumfang (Original) | Vorbereitung der Stamm lösungen für 10 Reaktions ansätze | Konzen tration der Stamm lösung | Volumen pro PCR-Ansatz | Volumen pro Negativ kontrolle | End- konzen- tration für 50 µl Reaktions- ansatz [8] |
| READYMIX enthält Taq- Plolymerase, Puffer, Magnesium- chlorid und Nukleotide | 500µl | verteilen auf 20 PCR Reaktionsgefäße, die vom Praktikanten zu kennzeichnen sind | 2x |
25µl |
25µl |
1,5u Taq Pol., 10mM Tris-HCl, 1,5mM MgCl2 0,2mM dNTP |
| Primer 1 (links) | 11,3nmol | alles gelöst in 227 µl A.dest., davon 22 µl + 22µl A.dest., verteilen auf 10 Reaktionsgefäße
Kennzeichen: P1 |
25pmol/µl |
2µl |
2µl |
1µM |
| Primer 2 (rechts) | 12,4nmol | alles gelöst in 248 µl A.dest., davon 22 µl + 22µl A.dest. verteilen auf 10 Reaktionsgefäße
Kennzeichen: P2 |
25pmol/µl |
2µl |
2µl |
1µM |
| A.dest.
(Ergänzung zu 50 µl Ansatz) |
19µl (20µl) |
21µl (20µl) |
||||
| Ziel DNA Bakteriophage Lambda | 1u =50µg | alles gelöst in 500 µl A.dest., davon 120 µl verteilen auf 10 Gefäße
Kennzeichen: L |
100 ng/µl |
2µl |
- |
200 ng pro 50 µl |
| Paraffinöl | mit der Tropfpipette je 2 große Tropfen (ca. 30 µl) auf 10 Gefäße verteilen
Kennzeichen: µl |
ca.2Tr. |
ca.2Tr. |
Tabelle 2: Reaktionsansatz für die PCR
5.2. Durchführung der PCR
Unmittelbar nach Zugabe von Taq-Polymerase und Paraffinöl wird die eigentliche PCR gestartet, indem die beiden Reaktionsgefäße in den schwimmfähigen Halterungen schnell in das Wasserbad ( 95 °C ) gegeben werden. Der Wechsel der Wasserbäder nach Tabelle 3 muss eingehalten werden. Am besten fertigt man sich vorher ein Protokoll an (vgl. Anlage). Die Spitze der Reaktionsgefäße mit dem Reaktionsgemisch muss voll in das Wasser eintauchen.
|
Temperatur |
Zeit |
Reaktionsschritt |
Zyklen |
|
95 °C |
2 min |
Denaturieren |
|
|
95 °C |
20 s |
Denaturieren |
|
|
62 °C |
30 s |
Hybridisieren |
25 x |
|
72 °C |
30 s |
Polymerisieren |
|
|
72 °C |
5 min |
Polymerisieren |
|
|
15 °C (wenn möglich 4°C, bzw. einfrieren) |
bis zur Gelelektrophorese |
Tabelle 3: Temperaturen und Zeiten der PCR
Alternativ kann die PCR in einem Thermocycler, der entsprechend dem Zeitplan programmiert wird, automatisch ablaufen.
5.3. Gelelektrophorese
Die Elektrophoresekammer wird nach Bedienungsanleitung mit 0,8%-igem Agarosegel in TBE-Puffer (Blue Genes: 1 %) gefüllt. Für das Beladen der Geltaschen werden folgende Lösungen zunächst hergestellt:
|
Spur 1 |
Spur 2 |
Spur 3 |
|
17 µl PCR-Ansatz (20 µl bei Lambda-Kit) |
17 µl Negativkontrolle (20 µl bei Lambda-Kit) |
8 µl Marker III |
|
- |
- |
8 µl A.dest. |
|
2 µl Auftragspuffer |
2 µl Auftragspuffer |
2 µl Auftragspuffer |
Tabelle 4: Beladungsschema für die Gelelektrophorese
Durchführung der Elektrophorese:
- Lambda-Kit: 90 min; 25 – 30 V
- Blue Genes Kit: 25 min; 10 – 120 V
In beiden Fällen soll der Farbstoff-Marker 2/3 der Gellänge erreicht haben.
5.4. Färben und Entfärben des Gels nach entsprechender Anleitung.
Immer mit Schutzhandschuhen !
Blue Genes - Kit:
- AzurB-Chlorid (1 ml ) mit 500 ml Wasser verdünnen; in eine Färbewanne geben.
- Gel vorsichtig in die Färbewanne gleiten lassen; darin muss es mindestens 15 min bleiben.
- Gel wird anschließend mehrmals mit entmineralisiertem Wasser entfärbt.
Nach 15 min sind die ersten Banden zu erkennen. Nach längerem Entfärben werden die Banden deutlicher.
Lambda-Kit:
- Elektroden entfernen, Pufferlösung abgießen.
- 1:1 (Aqua dest./ Färbelösungskonzentrat) verdünnte Färbelösung auf das Gel geben und 4 min dort belassen
- Färbelösung entfernen und dann mit 70 %-igem Spiritus 30 s lang überschichten.
- Spiritus verwerfen und dann mehrmals mit kaltem Leitungswasser übergießen.
Nach 10 min sind die ersten Banden sichtbar. Nach längerem Entfärben werden die Banden deutlicher.
6. Ergebnis und Diskussion
Die Abbildung zeigt ein gefärbtes Agarose-Gel mit dem Lambda Kit nach durchgeführter PCR mit 25 Zyklen

Bild 1: Ergebnis PCR
Auswertung:
Spur 1: PCR – Fragment mit 799 bp, amplifiziert aus 200 ng DNA
Spur 2: Bei der Negativkontrolle ist erwartungsgemäß keine Bande zu erkennen
Spur 3: Ungeschnittene Lambda-Phagen-DNA in hoher Konzentration (ca. 1 µg/ 20 µl) Wie zu erwarten ist, wird die sehr große DNA (48500 bp) nicht richtig getrennt, da die Porengröße des 0,8 %-igen Gels für diesen Größenbereich nicht geeignet ist.
Spur 4: Die Banden des DNA- Markers entsprechen denen in der Abbildung im Handbuch zum Blue Genes Kit. Sie können den einzelnen Fragmentgrößen leicht zugeordnet werden.
Zusammenfassung:
Wie die neuen Lehrpläne* zeigen, gewinnt auch das Thema Gentechnik und Biotechnik zukünftig in der Schule größere Bedeutung. Mit den erwähnten Experimentier-Kits ist es möglich, dass Schüler einfache molekularbiologische Versuche durchführen können wie z.B. das Schneiden von DNA mittels Restriktionsenzymen und der Nachweis von DNA-Fragmenten durch Gelelektrophorese. Im vorliegenden Artikel wurde die Experimentierpalette durch die PCR* erweitert. Die PCR kann exemplarisch mit einfachen Mitteln und relativ preiswert durchgeführt werden.
*) z.B. Baden-Württemberg, Niedersachsen, NRW, Sachsen
7. Bezugsquellen
(1) Blue Genes Kit: Fonds der Chemischen Industrie Karlstr. 21, 60329 Frankfurt
(2) Lambda Kit: National Centre for Biotechnology Education (NCBE), University of Reading, Whiteknights, Reading, RG6 2AJ, United Kingdom, German Version
(3) Komplettes Reagenzienset für den Genbaukasten, bestellbar bei Roche Diagnostic Boehringer Mannheim GmbH, Molekular Biochemicals D-68298 Mannheim Tel 0800/759 2226
(4) DNA-Molekulargewichtsmarker III, Artikelnr. 528552, Einzeln bestellbar als Reagenz des Genbaukastens unter dem Stichwort "Blue Genes" bei Roche Diagnostic Boehringer Mannheim GmbH, Molekular Biochemicals D-68298 Mannheim Tel 0800/759 2226
(5) Die beiden
Primer links (5‘-3‘, 24 bp): GAC GGT GCT GGG TAC GGA AGT GAA A
Primer rechts (5‘-3‘, 24 bp): GTT CCA TAC CGG CAT ACA GCG CAT C kann man z.B. bei der Fa. amersham pharmacia biotech GmbH, Postfach 5480, D-79021 Freiburg synthetisieren lassen. oligo.ordering@eu.apbiotech.com . Auch können die Primer bei der Fa. SIGMA ARK (Fax: 0695 007 0306; sigma-ark@eurnotes.sial.com )synthetisiert werden.
Primer Design: Die Primer wurden mit dem über das Internet frei zugänglichen Programm konstruiert: (Primer 3), Center for Genome Research at the Whitehead Institute for Biomedical Research in Cambridge, Massachusetts, USA: http://www.genome.wi.mit.edu/ .Die Nukleotidsequenz des Lambda-Phagen ist über das Internet zu erhalten von: National Center for Biotechnology Information National Library of MedicineBuilding 38A, Room 8N805 Bethesda, MD 20894 http://www.ncbi.nlm.nih.gov/
(6) z.B. Core-System der Promega GmbH, High Tech Park, Schildkrötstr. 15, 68199 Mannheim oder entsprechendes Taq-DNA-Polymerase-Kit und dNTP-Mischung der Fa.Eppendorf GmbH, 22331 Hamburg auch bei Eppendorf vgl. (10)
(7) Es gibt sehr viele Anbieter: z.B. BIO-RAD laboratories GmbH, Postfach 450133, 80901 München, Biometra GmbH, Rudolf Wissell Str. 30, D-37079 Göttingen, Eppendorf-Netheler-Hinz GmbH, Friedensstr.116, 51145 Köln
(8) z.B. Multiply-Pro, 500 m l, Best.Nr. 72.735, 500 Stck./Beutel,Preis ca. 60.- . Bestellbar bei Sarstedt AG & Co. , Postfach 1220, D-51582 Nümbrecht
( 9) Paraffinöl (mineral oil) Best.Nr. 69794, 100 ml, Preis ca. 20.-, Fluka Sigma-Aldrich Chemie GmbH Grünwaldweg 3, 82041 Deisenhofen
(10) Eppendorf-Netheler-Hinz GmbH, Friedensstr.116, 51145 Köln
(13) Promega GmbH, High Tech Park, Schildkrötstr. .15, 68199 Mannheim
(14) Sigma-Aldrich GmbH, Eschenstr. 5, 82024 Taufkirchen
(15) Amersham Pharmacia Biotech GmbH, Postfach 5480, 79021 Freiburg
(16) Sigma Ark (Fax: 0695 007 0306; sigma-ark@eurnotes.sial.com )
8. Literatur
[ 1] H.BAYRHUBER – U.KULL: Linder Biologie.- 21. Aufl.- Hannover: Schroedel Verlag, 1998
[ 2] H.G. GASSEN – G.E. SACHSE – A. SCHULTE: PCR. – Stuttgart: Gustav Fischer, 1994
[ 3] TH. DINGERMANN – I. ZÜNDORF: Gentechnik Biotechnik. – Stuttgart: Wissenschaftliche Verlagsgesellschaft 1999
[ 4] T. STRACHAN – A.P. READ: Molekulare Humangenetik - Heidelberg: Spektrum Akademischer Verlag 1996
[ 5] M. BENECKE – Kriminalbilologie. – Bergisch Gladbach: BLT 1999
[ 6] H. BAYRHUBER – E.R. LUCIUS: Handbuch der praktischen Mikrobiologie. Band 1. – Hannover: Metzler Schulbuchverlag 1992
[ 7] T.A. BROWN: Gentechnologie für Einsteiger.- Heidelberg: Spektrum Akademischer Verlag 1996
[ 8] PROMEGA: Technical Bulletin, Instructions for use of products M7660 and M7665. – Download Adresse: www.promega.com
9. Kostenübersicht
Kostenübersicht für die PCR (ca. in DEM ohne MWSt und Transport)
Zusätzlich zum vorhandenen Lambda-Kit bzw. Blue Genes Kit
| Komponente | Liefermenge | ausreichend für wieviel PCR | Preis pro Lieferung | Verbrauch für 10 PCR | Preis für 10 PCR | Preis pro PCR |
| Ready Mix enthält Taq-Polymerase Puffer, MgCl2, Nukleotide (13)(14) | 500 µl | 20 | 30,40 | 250 µl | 15,2 | 1,52 |
| Lambda Phagen DNA Lyophilized (14) | 1 unit
(ca. 50 µg ) |
250 | 35,00 | 2 µg | 1,40 | 0,14 |
| Primer (15)(16) | 11,3 nmol | 250 | 100 | 20 µl | 4,00 | 0,4 |
| PCR-Gefääße 0,5ml
Multiply Pro (8) |
500 | 60.00 | 1,20 | 0,12 | ||
| 225,00 | 2,1 |
(13) Promega GmbH, High Tech Park, Schildkrötstr. .15, 68199 Mannheim
(14) Sigma-Aldrich GmbH, Eschenstr. 5, 82024 Taufkirchen
(15) Amersham Pharmacia Biotech GmbH, Postfach 5480, 79021 Freiburg
(16) Sigma Ark (Fax: 0695 007 0306; sigma-ark@eurnotes.sial.com )
10. Zeitprotokoll
| Zyklus | min |
s |
Zyklus | min |
s |
|||||||
| Start |
°C |
0 |
0 |
ok |
°C |
ok |
||||||
|
95 |
bis |
2 |
0 |
|||||||||
|
1 |
95 |
bis |
2 |
20 |
14 |
95 |
bis |
19 |
40 |
|||
|
62 |
bis |
2 |
50 |
62 |
bis |
20 |
10 |
|||||
|
72 |
bis |
3 |
20 |
72 |
bis |
20 |
40 |
|||||
|
2 |
95 |
bis |
3 |
40 |
15 |
95 |
bis |
21 |
0 |
|||
|
62 |
bis |
4 |
10 |
62 |
bis |
21 |
30 |
|||||
|
72 |
bis |
4 |
40 |
72 |
bis |
22 |
0 |
|||||
|
3 |
95 |
bis |
5 |
0 |
16 |
95 |
bis |
22 |
20 |
|||
|
62 |
bis |
5 |
30 |
62 |
bis |
22 |
50 |
|||||
|
72 |
bis |
6 |
0 |
72 |
bis |
23 |
20 |
|||||
|
4 |
95 |
bis |
6 |
20 |
17 |
95 |
bis |
23 |
40 |
|||
|
62 |
bis |
6 |
50 |
62 |
bis |
24 |
10 |
|||||
|
72 |
bis |
7 |
20 |
72 |
bis |
24 |
40 |
|||||
|
5 |
95 |
bis |
7 |
40 |
18 |
95 |
bis |
25 |
0 |
|||
|
62 |
bis |
8 |
10 |
62 |
bis |
25 |
30 |
|||||
|
72 |
bis |
8 |
40 |
72 |
bis |
26 |
0 |
|||||
|
6 |
95 |
bis |
9 |
0 |
19 |
95 |
bis |
26 |
20 |
|||
|
62 |
bis |
9 |
30 |
62 |
bis |
26 |
50 |
|||||
|
72 |
bis |
10 |
0 |
72 |
bis |
27 |
20 |
|||||
|
7 |
95 |
bis |
10 |
20 |
20 |
95 |
bis |
27 |
40 |
|||
|
62 |
bis |
10 |
50 |
62 |
bis |
28 |
10 |
|||||
|
72 |
bis |
11 |
20 |
72 |
bis |
28 |
40 |
|||||
|
8 |
95 |
bis |
11 |
40 |
21 |
95 |
bis |
29 |
0 |
|||
|
62 |
bis |
12 |
10 |
62 |
bis |
29 |
30 |
|||||
|
72 |
bis |
12 |
40 |
72 |
bis |
30 |
0 |
|||||
|
9 |
95 |
bis |
13 |
0 |
22 |
95 |
bis |
30 |
20 |
|||
|
62 |
bis |
13 |
30 |
62 |
bis |
30 |
50 |
|||||
|
72 |
bis |
14 |
0 |
72 |
bis |
31 |
20 |
|||||
|
10 |
95 |
bis |
14 |
20 |
23 |
95 |
bis |
31 |
40 |
|||
|
62 |
bis |
14 |
50 |
62 |
bis |
32 |
10 |
|||||
|
72 |
bis |
15 |
20 |
72 |
bis |
32 |
40 |
|||||
|
11 |
95 |
bis |
15 |
40 |
24 |
95 |
bis |
33 |
0 |
|||
|
62 |
bis |
16 |
10 |
62 |
bis |
33 |
30 |
|||||
|
72 |
bis |
16 |
40 |
72 |
bis |
34 |
0 |
|||||
|
12 |
95 |
bis |
17 |
0 |
25 |
95 |
bis |
34 |
20 |
|||
|
62 |
bis |
17 |
30 |
62 |
bis |
34 |
50 |
|||||
|
72 |
bis |
18 |
0 |
72 |
bis |
35 |
20 |
|||||
|
13 |
95 |
bis |
18 |
20 |
Ende |
72 |
bis |
40 |
20 |
|||
|
62 |
bis |
18 |
50 |
|||||||||
|
72 |
bis |
19 |
20 |



