GeneControl
(Mechanismen der Genregulation nach Jacob-Monod)
Die Kontrolle der Transkription ist der wichtigste Mechanismus, mit dem
Organismen ihre Genexpression und damit ihr Proteinrepertoire in der Zelle
variieren. Bei einer Vielzahl prokaryotischer Operons geschieht dies durch
negativ- und/oder positiv wirkende regulatorische Proteine. Die Aktivität der
regulatorischen Proteine hängt vom physiologischen Zustand der Zelle und dieser
wiederum von der Umwelt (Nahrungsquellen, Temperatur etc.) ab. Im allgemeinen
reagieren regulatorische Systeme bereits auf kleine Änderungen der Umwelt und
passen die Zelle jeweils optimal an die vorherrschenden Bedingungen an. Als
Genexpression bezeichnet man den Vorgang, bei dem eine bestimmte DNA-Sequenz
zuerst in RNA und anschließend in Proteinmoleküle, den Genprodukten, übersetzt
wird.
Ein Organismus kann seine Gene entweder immer (konstitutiv) oder nur
unter bestimmten physiologischen Bedingungen (reguliert) exprimieren. Von
seltene Proteinen findet man oft nur 10, von anderen Proteinen mehr als 105 Kopien
in der Zelle vor. Ein vielzelliger Organismus enthält zwar dasselbe Genom, die
verschiedenen Zelltypen können aber einen unterschiedlichen Satz von Proteinen
synthetisieren, der zu stark abweichenden Morphologien und Funktionen führen
kann (z. B. Nervenzellen und Blutzellen).
Aufbau eines prokaryotischen Operons
Eine prokaryotische Transkriptionseinheit, ein Operon, besteht im
einfachsten Fall aus einer DNA Sequenz, die eine Bindungsstelle für die
RNA Polymerase, den Promotor, eines oder mehrere Strukturgene sowie
einem Signal zur Termination der Transkription, den Terminator,
enthält. Promotoren enthalten zusätzlich häufig Bindungsstellen für
regulatorische Proteine,
Aktivatoren und Repressoren, die Aktivität der RNA Polymerase
kontrollieren. Die Bindungsstelle eines Repressors auf der DNA nennt
man
Operator. mRNAs, die kodierende Regionen mehrerer Gene enthalten, aber
von einem Promotor aus transkribiert werden, bezeichnet man als
polycistronisch oder polygen. Sie sind charakteristisch für Prokaryoten.
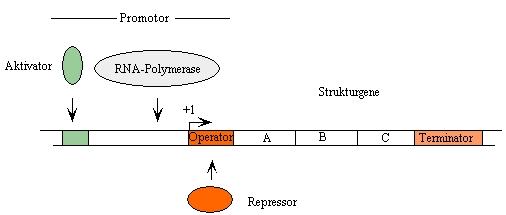
Abb.1:
Struktur und Funktionskomponenten eines prokaryotischen Operons. Mit
+1 wird der Transkriptionsstart, also das erste Nukleotid einer neu
synthetisierten RNA, bezeichnet Weitere Erklärungen im Text.
Prinzipien der Regulation prokaryotischer Operons.
Bevor wir zur Beschreibung ausgewählter Operons und deren
Regulationsmechanismus aus E. coli kommen, ist es wichtig, einige Begriffe zu
definieren: Man unterscheidet bei regulierbaren Operons zwischen induzierbaren
und reprimierbaren Systemen. Je nachdem, ob an der Regulation eines Promotors
ein Repressor- oder ein Aktivatorprotein beteiligt ist spricht man auch von
negativer oder positiver Kontrolle.
Definition der Induktion: Die Bindung eines (meist kleinen) Moleküls, das als
"Effektor" oder "Kofaktor" bezeichnet wird, führt zur Erhöhung der
Transkriptionsrate. Induzierbare Systeme findet man bei katabolen (abbauenden)Stoffwechselprozessen.
Ein Beispiel für ein induzierbares Systeme unter Beteiligung eines Repressors
stellt das Laktose (lac)-Operon dar. Das regulatorische Protein ist der Laktose
Repressor (LacR), der Effektor ist der Zucker Laktose. Das Arabinose (ara)-Operon
hingegen ist ein Beispiel für ein induzierbares System unter Beteiligung eines
Aktivators, hier AraC. Der Kofaktor ist der Zucker Arabinose.
Definition der Repression: Die Bindung eines (meist kleinen) Moleküls, das
als "Effektor" oder "Kofaktor" bezeichnet wird, führt zur Verringerung der
Transkriptionsrate. Reprimierbare Systeme findet man bei anabolen (aufbauenden)
Stoffwechselprozessen, für die häufig eine Endprodukthemmung charakteristisch
ist. Beispiele sind das Tryptophan (trp)-Operon (bestehend aus dem
Tryptophan-Repressor und dem Effektor Tryptophan), das für die Synthese von
Tryptophan zuständig ist oder das fad-Operon, das für die Synthese kurzkettiger
Fettsäuren (diese sind gleichzeitig der Effektor) zuständig ist. Das
Regulatorprotein heißt FadR.
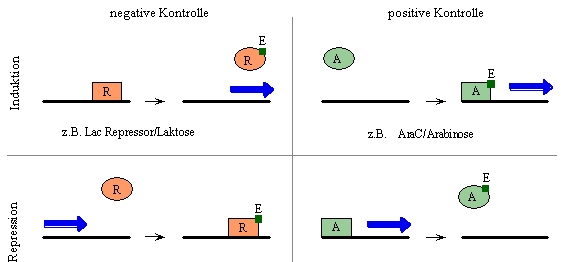
Abb.2 Induzierbare und Reprimierbare Systeme. R: Repressor, A: Aktivator, E: Effektor
Aufbau und Funktion prokaryotischer Promotoren
Da die RNA-Synthese nicht irgendwo auf der DNA beginnen sollte, sondern genau
vor einer kodierenden Sequenz, erfolgt die Bindung der RNA-Polymerasen über eine
spezifische DNA-Sequenz, die stromaufwärts (upstream) eines kodierenden
DNA-Abschnitts liegt, dem Promotor. Durch Vergleich von über 250
Promotorsequenzen konnte gezeigt werden, dass prokaryotische Promotoren einige
konservierte Sequenzmotive aufweisen, die auch als Konsensussequenzen bezeichnet
werden. Eine davon liegt 10 Basenpaare stromaufwärts vom Transkriptionsstart
(dieser wird als Position +1 auf der DNA bezeichnet). Die Konsensussequenz
dieser als "10 Region oder Pribnow Box bezeichneten Nukleotidfolge lautet
5´-TATAAT-3´. Ca. 35 Basenpaare stromaufwärts gibt es eine zweite Folge von
konservierten Nukleotiden, die "35 Region. Diese hat die Konsensussequenz
5´-TTGACA-3´. Interessanterweise wurde bisher kein Promotor gefunden, der eine
perfekte Konsensus-Sequenz aufweist. Der Abstand zwischen dem
Transkriptionsstart und dem ersten Nukleotid der "10 Region beträgt 5 - 8
Basenpaare, der Abstand zwischen der "10 Region und der " 35 Region 16 - 18
Basenpaare. Die Sequenz zwischen der -35 Region und dem Transkriptionsstart
bezeichnet man auch als Promotor Kernregion. Die flankierenden Regionen
stromaufwärts bzw. stromabwärts der Kern-Region sind häufig Bindungsstellen von Aktivatoren und Repressoren.
Aktivatoren oder Repressoren können abhängig vom physiologischen Zustand der
Zelle die Expression der Gene vom betreffenden Promotor aus kontrollieren.
Mittlerweile kennt man ca. 150 - 200 verschiedenen Regulatorproteine, deren
Wirkungsweise zum Teil gemeinsame Prinzipien unterliegen. In E. coli werden mehr
als 130 Promotoren durch negative oder positive regulatorische Proteine
kontrolliert. Die Mehrzahl der Repressormoleküle reprimiert die Transkription,
indem sie den Zugang der RNA-Polymerase zum Promotor sterisch blockieren. Um
dies zu gewährleisten, muss ein Repressor an eine DNA Sequenz, den Operator, in
unmittelbarer Nähe des Promotors binden. Im Gegensatz zu Repressoren binden Aktivatormoleküle fast ausschließlich
stromaufwärts ("upstream") von der -35 Region mit variablem Abstand zum
Promotor. Es konnte gezeigt werden, dass der Aktivierung ein Kontakt zwischen
dem Aktivator und der RNA-Polymerase zugrunde liegt.
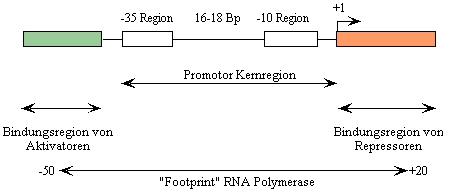
Abb.3:
Prinzipieller Aufbau eines prokaryotischen Promotors. Nicht alle
Promotoren werden durch Aktivatoren und/oder Repressoren kontrolliert und haben
demzufolge auch keine Bindungsstellen für diese Regulatorproteine. Die Lage der
Bindungsstellen für Regulatorproteine können zwischen verschiedenen Promotoren
variieren. "+1" bezeichnet den Transkriptionsstart, d. h. das erste Nukleotid
in einer neu synthetisierten RNA. Die anderen Positionen auf der DNA werden
relativ zu diesem Punkt definiert und zwar stromabwärts mit "+2", "+3" etc. und
stromaufwärts mit "-1", "-2" etc..
Eine Promotorsequenz liefert Informationen, die den Transkriptionsstartpunkt sowie die Promotorstärke, festlegen. Die Promotorstärke, die sich bei E. coli Promotoren um mehr als 1000 fach unterscheiden kann, wird definiert als die Rate, mit der eine RNA-Polymerase einen Promotor produktiv, d.h. unter Bildung einer funktionsfähigen RNA, verlässt. Die stärksten E. coli Promotoren sind diejenigen, die zur Synthese der ribosomalen RNAs (Prrn) gebraucht werden. Sie können den gesamten Prozess der Transkriptionsinitiation etwa jede Sekunde durchlaufen. Im Gegensatz dazu gibt es Promotoren, die nur sehr selten transkribiert werden, da ihr Genprodukt nur in geringen in der Zelle gebraucht wird. Schließlich gibt es viele Promotoren, wie z. B. die Promotoren Plac oder Para die nur bei Bedarf angeschaltet werden. Die Programme nach denen diese Promotoren jeweils funktionieren sind verschlüsselt in der Promotorsequenz. Wie sich die Änderung der DNA-Sequenz in der -10 bzw. -35 Region auf die Stärke eines Promotors auswirkt, werden Sie in diesem Experiment messen.
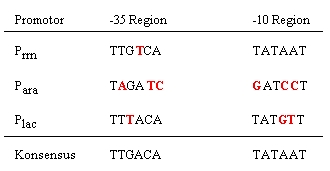
Abb.4 Sequenzen der -10 und -35 Regionen ausgewählter E. coli Promotoren. Die Positionen, an denen die Sequenzen von der Konsensussequenz abweichen, sind durch Fettdruck hervorgehoben.
Die E. coli RNA-Polymerase
Das Schlüsselenzym der Gen-Expression ist die
DNA-abhängige RNA-Polymerase, die zur RNA-Synthese die DNA als Matrize
verwendet. E. coli verfügt im Gegensatz zu höheren Eukaryoten nur über eine
Spezies der DNA-abhängigen RNA-Polymerase, die für die Synthese aller RNA
Moleküle in der Zelle verantwortlich ist. E. coli Zellen besitzen ca. 3000
RNA-Polymerase Moleküle, die permanent mit der Synthese von bis zu 1600 RNA
Molekülen, darunter 400-800 verschiedene messenger-RNAs, 700 Vorläufermolekülen
für ribosomale-RNAs und ca. 100 unterschiedlichen transfer-RNAs beschäftigt
sind.
RNA-Polymerasen bestehen aus mehreren Untereinheiten, die
unterschiedliche Funktionen ausüben. Die bakterielle RNA-Polymerase besteht aus
5 Untereinheiten, nämlich 2 alpha, beta, beta´, und einer s Untereinheit.
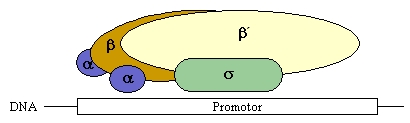
Abb.5 Aufbau und Funktion der E. coli RNA-Polymerase. Die β-Untereinheit ist das katalytische Zentrum der RNA-Synthese. Über die β-Untereinheit (Sigmafaktor) erkennt und bindet eine RNA-Polymerase einen Promotor. Die β- Untereinheit ist zudem die Kontaktstelle für viele Aktivatorproteine.
Die ribosomalen RNA (rrn) Operons von E. coli
Der bakterielle RNA-Pool besteht hauptsächlich aus drei
Klassen von RNAs: ribosomale RNA (rRNA, ca. 90%), transfer RNA (tRNA, ca. 7-8%)
und mRNA (mRNA, ca. 2-3%). Es gibt drei verschiedene Typen von rRNAs, nämlich
die 5S, die 16S und die 23S rRNA. In einem Operon werden alle drei rRNA Gene in
Form einer einzigen Vorläufer-RNA synthetisiert und anschließend von RNasen in
die drei Einzel-RNAs gespalten. Eine Bakterienzelle enthält ca. 45 000 rRNA, die
zusammen mit etwa 50 Proteinen die 15 000 Ribosomen der Zelle bilden. Die
benötigte Menge an rRNA in einer Zelle ist so hoch, dass sieben verschiedene
rRNA Operons mit der Synthese der rRNAs beschäftigt sind. Die Promotoren, von
denen die rRNA Gene transkribiert werden, werden als Prrn bezeichnet Da rRNAs in
einer sich schnell teilenden Bakterienzelle ständig benötigt werden, werden die
Promotoren nicht durch regulatorische Proteine kontrolliert. Man sagt, die
Promotoren werden konstitutiv exprimiert.
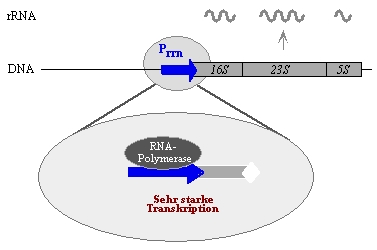
Abb.6 Aufbau eines rRNA Operons aus E. coli. Die drei ribosomalen RNAs werden in Form einer einzelnen rRNA synthetisiert und nach der Synthese prozessiert. Der Pfeil symbolisiert den Promotor Prrn.
Das Laktose (lac) Operon von E. coli.
Die Gene des Laktose (lac) Operons (lacZ, lacY, lacA) von
E. coli ermöglichen dem Bakterium, Laktose als Kohlenstoffquelle zu nutzen. Die
Expression dieser Gene wird durch den Promotor Plac kontrolliert. Befindet sich
keine Laktose im Wachstumsmedium, so wird der Promotor durch den Lac-Repressor
(kodiert durch das laci Gen) reprimiert Dies geschieht durch die Bindung des Lac
Repressors an den lac Operator, einer DNA-Sequenz in der Lac-Promotorregion.
Durch die Bindung des Lac Repressors wird die RNA Polymerase an der Bindung des
Promotors gehindert und damit die Transkription verhindert.
Steht Laktose als Kohlenstoffquelle zur Verfügung, so
bindet der Lac-Repressor Laktose und verändert dadurch seine dreidimensionale
Gestalt, mit der Folge, seine Bindungsfähigkeit an den lac Operator verloren
geht. Die RNA-Polymerase kann deshalb an die Promotorregion binden und die Gene
für den Laktose-Abbau transkribieren.
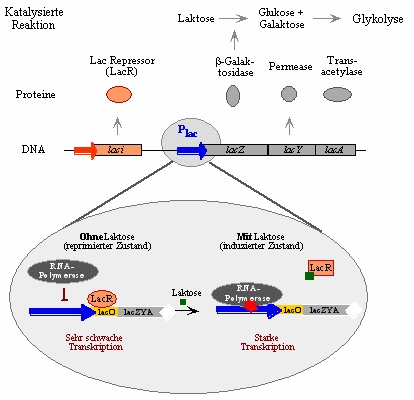
Abb.7 Aufbau und Funktion des E. coli Laktose (lac) Operons. Die Funktion der Transacetylase ist nicht geklärt. Die Pfeile symbolisieren Promotoren. Nähere Erläuterungen finden sich im Text.
Das Arabinose (ara) Operon von E. coli
Das Arabinose (ara) Operon aus E. coli enthält Gene (ara
A, araB und araD), die für den Abbau des Zuckers Arabinose in der Zelle
zuständig sind. Die Gene werden durch den Promotor Para reguliert. Dieser
Promotor wird nur dann aktiviert, wenn Arabinose im Nährmedium vorhanden ist.
Dies geschieht durch das Aktivatorprotein AraC (kodiert durch das araC Gen),
dessen Aktivität durch Arabinose gesteuert werden kann: AraC ist inaktiv, wenn
es keine Arabinose gebunden hat, d.h. es kann die Transkription des
PBADPromotors nicht aktivieren. Erst nach Bindung an Arabinose kann es seine
DNA-Erkennungssequenz (araI) binden und durch einen direkten Kontakt mit der RNA
Polymerase deren Transkriptionshäufigkeit erhöhen. Das Resultat ist die
verstärkte Expression der Gene, die für den Arabinose-Abbau zuständig sind.
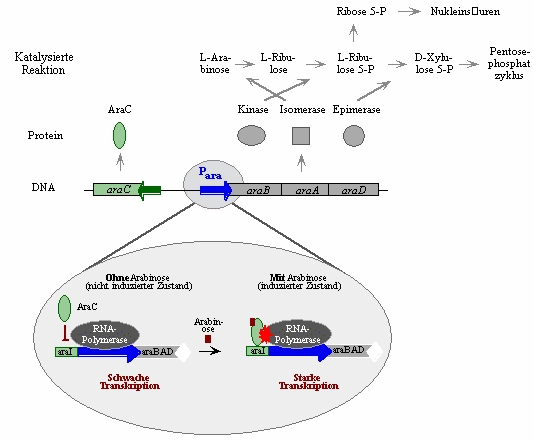
Abb.8 Aufbau und Funktion des E. coli Arabinose (ara) Operons. Die Pfeile symbolisieren Promotoren. Nähere Erläuterungen finden sich im Text.
Experimentieranleitung
Im Experiment soll die Veränderung der Genaktivität der
drei in der Einleitung erwähnten Promotoren, Plac, Para und Prrn als Antwort auf
einen Wechsel der Nahrungsbedingungen im Wachstumsmedium gemessen werden. Wie
bereits erwähnt, wird einer dieser drei Promotoren (Plac) durch den Laktose
Repressor kontrolliert, eine weiterer (Para) durch das AraC Protein. Der dritte
Promotor (Prrn) wird nicht durch ein regulatorisches Protein kontrolliert, man
sagt, er wird konstitutiv exprimiert.
Die Identifizierung und Charakterisierung der Promotoren soll durch das Experiment erfolgen.



