PhageRestriktion
(Mikrobiologischer Nachweis von Restriktionsenzymen in E. coli)
Der Versuch reproduziert das klassische Experiment, aus dem Arber, Nathans und Smith Ende der 60er Jahre die Existenz der heute wohlbekannten Restriktionsenzyme sowie der zugehörigen Schutzenzyme, der Methyltransferasen, postuliert und später auch nachgewiesen haben. Ausgangspunkt für das Experiment sind die E. coli Stämme a bzw. aR. Letzterer trägt zusätzlich Gene, die für ein Restriktionsenzym (R) sowie die dazugehörige Methyltransferase M-EcoR1 kodieren (Abb.1).
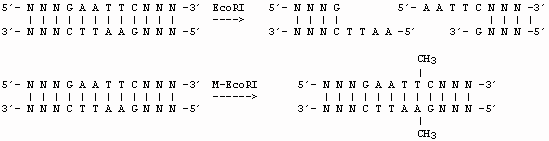
Abb.1: Reaktionsmechanismen des Restriktionsenzyms EcoRI und der EcoRI Methyltransferase. Das Restriktionsenzym spaltet die DNA-Sequenz an der Erkennungsstelle 5´-GAATTC-3´und lässt sog. "klebrige Enden" (sticky ends) zurück. Die Methyltransferase überträgt einen Methylrest auf eine Base in je einem Strang der Erkennungssequenz.
Beide Stämme werden mit Lambda-Phagen infiziert, die aus dem Stamm a stammen,
deren DNA also nicht methyliert ist. Um die Herkunft der Phagen deutlich zu
machen, werden diese als l.a bezeichnet.Das Infektionsvermögen der Phagen auf den beiden Stämmen wird anhand der
Anzahl der gebildeten Plaques auf den Bakterienrasen bestimmt und als
"Plattierungseffizienz" (engl. "efficiency of plating", EOP) bezeichnet.
Diese ist bei Stamm aR ca.1000 - 2000 fach geringer als bei a (vgl. die
Plattierungseffizienz von l.a auf a und aR). Hieraus kann man schließen, dass aR
ein Schutzsystem besitzt, das den Stamm effizient vor einer Infektion durch l.a
schützt. Allerdings gelingt es einigen Phagen, das Schutzsystem zu umgehen.
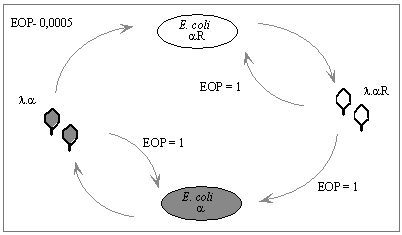
Abb.2:
Wirtszellkontrollierte Restriktion und Modifikation des Phagen Lambda.
EOP=Efficiency of Plating (Plattierungseffizienz), angegeben in relativen
Werten.
Die Nachkommen dieser Phagen (l.aR) wiederum können dann ihren Wirtstamm mit der gleichen Effizienz infizieren wie den Stamm a (Abb.2). Dies liegt daran, dass die DNA der aus aR freigesetzten Phagen (l.aR) durch das Enzym Methyltransferase an den Erkennungsstellen des Restriktionsenzyms methyliert wurde und somit von aR nicht mehr als fremd erkannt werden kann. Die eigentliche Funktion der Methyltransferase ist es, durch Methylierung der bakteriellen DNA diese vor dem Abbau durch die eigenen Restriktionsendonukleasen zu schützen. Infiziert und lysiert der Phage l.aR erneut den Stamm E.coli a (der kein Restriktions- und Methylierungssystem besitzt), wird die neu synthetisierte Phagen-DNA nicht methyliert und kann von aR wiederum als fremd erkannt werden. Damit schließt sich der im Bild dargestellte Zyklus. Restriktionsendonukleasen werden aufgrund ihrer Struktur und Wirkungsweise in 3 Klassen (Typ I bis III) eingeteilt. Sind Restriktionsenzyme des Typs I und III durchaus von wissenschaftlichem Interesse, so spielen sie jedoch in der Gentechnik so gut wie keine Rolle, da ihre Erkennungsstelle auf der DNA nicht identisch mit der Schnittstelle ist, und somit keine DNA-Fragmente voraussagbarer Länge entstehen. Wenn man von Restriktionsenzymen im Zusammenhang mit Gentechnik spricht, so meint man deshalb ausschließlich Restriktionsendonukleasen des Typs II. Dies sind Enzyme, die 4-, 5-, 6 oder 8 Basenpaare lange DNA-Sequenzen erkennen und dort beide Stränge der DNA schneiden. Dabei entstehen 5´-phosphorylierte und 3´-hydroxylierte Enden, die entweder einzelsträngig und überhängend ("sticky ends") oder doppelsträngig und glatt ("blunt ends") sind. Auf diese Weise ist es möglich DNA-Moleküle sequenzspezifisch zu fragmentieren (man sagt auch "verdauen").

Abb.3: Eigenschaften von Restriktionsenzymen
Der Name eines Restriktionsenzyms besteht aus drei Buchstaben, die den
Wirtsorganismus, aus dem das Enzym stammt, deutlich machen (z. B. "Eco"
für
Escherichia coli oder "Hin" für Haemophilus influenzae). Ein weiterer
Buchstabe steht für die genauere Charakterisierung des Wirtsstammes,
etwa durch die Beschreibung seines Serotyps. Weist ein Wirtsstamm
mehrere Restriktions- und Modifikationssysteme auf, so werden diese mit
römischen Zahlen gekennzeichnet. Derzeit sind mehr als 2000
Restriktionsenzyme bekannt, mehr als 500 davon kommerziell erhältlich.
Viele Restriktionsenzyme werden heute aus Kostengründen nicht mehr aus
ihrem natürlichen Wirt isoliert, sondern rekombinant in E. coli erzeugt.

Tab.1 Erkennungssequenzen einiger Restriktionsendonukleasen. Die mit "*" gekennzeichnete Base wird durch die zugehörige Methyltransferase methyliert. BamHI und Sau3AI erzeugen trotz unterschiedlicher Erkennungssequenzen kompatible Überhänge. An der dritten Position der Erkennungsstelle von EcoRII kann entweder ein A oder ein T stehen. "/" bezeichnet die Spaltstelle des Enzyms. "N" steht für eine beliebige Base.



