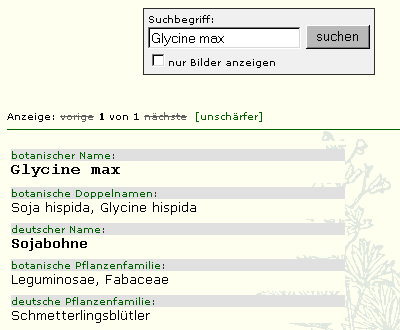
Sequenzanalyse im Internet
1. Einführung
Heutzutage gibt es frei zugängliche Datenbanken mit Nukleotid- und Aminosäuresequenzen im Internet, die Wissenschaftler analysiert und hier eingespeist haben. Beim
können Sie in den Beschreibungen der Datensätze von solchen Sequenzen im Volltext nach Wörtern suchen. Dabei sind allein in der "hauseigenen" GenBank schon über 13 Milliarden Basen aus über 100 000 biologischen Arten gespeichert.
Porine (=Porenproteine) sind Membranproteine, die die Diffusion von einzelnen Stoffen durch Biomembranen spezifisch erleichtern. In Tieren muss z.B. der Energieträger Glucose von einer Zellsorte in das Blutkreislaufsystem ausgeschleust werden und von anderen Zellen von daher importiert werden. Der Wasserhaushalt höherer Organismen wird aktiv durch die Klasse der Aquaporine reguliert, wobei in Menschen mehr als 150 Liter pro Tag bewegt weden.
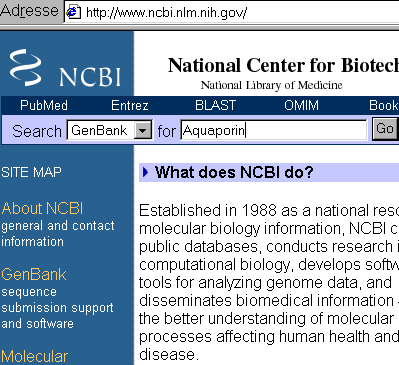
2. Aufgabe
Geben Sie als Suchwort Aquaporin ein.
Klicken Sie auf Go.
Sie erhalten kurz beschriebene Hyperlinks von Datensätzen, in deren Beschreibung das Wort Aquaporin vorkommt:
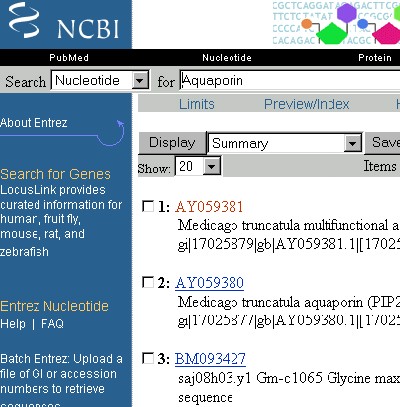
Wenn man die Identifikationsnummer der Sequenz (hier lautet die erste AY059381) anklickt, erhält man den ganzen Datensatz.
3. Beispiel
Es wurde der dritte Datensatz angeklickt: eine Nukleotidsequenz aus Glycine max, der Sojabohne:
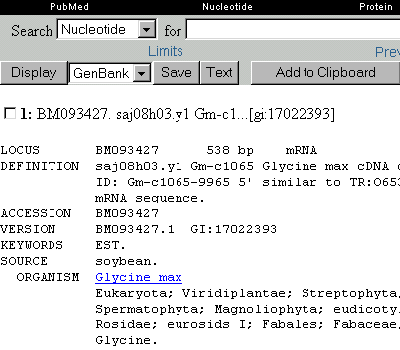
- LOCUS: gibt die Anzahl der Basenpaare (bp=basepairs) und den Typ der Nukleinsäure an.
- SOURCE: gibt den englischen Artnamen an.
- ORGANISM: gibt den lateinischen Artnamen und die systematischen
Einheiten an, zu denen der Organismus zugeordnet ist.
4. Aufgabe
Wählen Sie einen Datensatz, der nicht zur Sojabohne gehört.
Finden Sie heraus, in welchem Organismus die Nukleotidsequenz gefunden
wurde.
Lateinischer Artname: ________________________________________________
Deutscher Artname: __________________________________________________
5. Hilfe
Falls Sie den englischen Namen nicht übersetzen können, finden Sie anhand des lateinischen Namens den deutschen Artnamen mit dem Internet heraus, z.B. mit Floraweb von der Linksammlung aus:
6. Beispiel
7. Die Nukleotidsequenz
Die Einzelbasen werden unter BASE COUNT (weiter unten auf der Seite mit der Nukleotidsequenz) aufsummiert. Dann folgt die Nukleotidsequenz.
8. Beispiel

Das Bild ist rechts abgeschnitten. Am Beginn einer jeden Zeile wird die Nummer der ersten Base dieser Zeile angegeben.
9. Aufgabe
Finden Sie die Anzahlen der Basen auf der von Ihnen gewählten
Nukleotidsequenz heraus.
Adenin |
________ |
Cytosin |
________ |
Guanin |
________ |
Thymin |
________ |
10. Speichern von Nukleotidsequenzen
Speichern Sie jetzt drei Nukleotidsequenzen.
Gehen Sie dazu zurück auf die Suchergebnisse. Wählen Sie dazu ein anderes Format: FASTA. Klicken Sie auf Display.
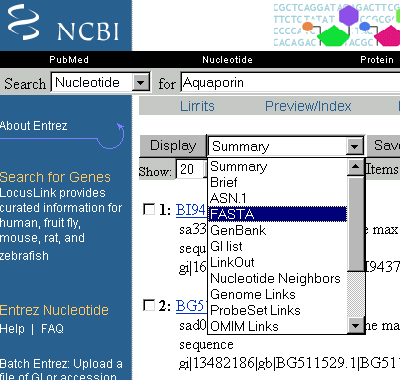
Markieren Sie mit einem Klick (Häkchen setzen links neben die Nummer) die erste Sequenz, die Sie speichern wollen. Klicken Sie dann auf Save. Speichern Sie die Datei unter einem sprechenden Namen.
Machen Sie das Gleiche mit den anderen zwei Sequenzen.
Die Dateien können Sie später in WORD laden und ausdrucken.
11. Vergleich der Nukleotidsequenz
Jetzt werden Sie die von Ihnen gefundene Nukleotidsequenz mit anderen Nukleotidsequenzen aus den NCBI-Datenbanken vergleichen, um ähnliche Sequenzen, die für ähnliche Proteine codieren, zu finden.
12. Aufgabe
Öffnen Sie die erste Nukleotidsequenz in WORD und kopieren Sie sie, indem Sie sie mit der Maus markieren und (im Menü von WORD) mit Bearbeiten/Kopieren in die Zwischenablage befördern.
Gehen Sie dann auf die Startseite der NCBI zurück und klicken Sie auf BLAST.
Auf der aufgerufenen Seite wählen Sie unter Nucleotide BLAST den Menüpunkt Standard nucleotide-nucleotide BLAST [blastn].
Klicken Sie dort in das Feld Search und fügen durch (im Browsermenü) Bearbeiten/Einfügen die Nucleotidsequenz ein.
Standardmäßig wird in den Datenbanken GenBank des NCBI, EMBL (Großbritannien), DDBJ (Japan) und PDB (Protein Database Brookhaven, USA) gesucht (im Dropdownmenü Choose Database der voreingestellte Eintrag nr.).
Klicken Sie auf den Button Blast.
Auf der folgenden Seite klicken Sie auf Format, um eine formatierte Ausgabe zu erhalten.
13. Erklärung
Oben auf der folgenden Seite sehen Sie die Übereinstimmungen in grafischer Form:
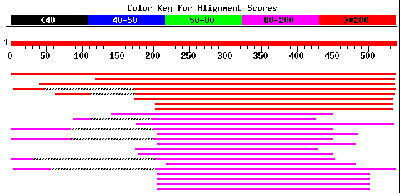
Der dicke rote Balken mit der Basenskala entspricht der eingegebenen Sequenz.
Darunter sind die Sequenzen als dünnere farbige Striche grafisch dargestellt, die teilweise Übereinstimmung zeigen.
Farblich wird das Maß der Übereinstimmung in Anzahl Basen angezeigt:
![]()
![]()
Die Länge und Lage der Striche zeigt die ungefähre Übereinstimmung.
Eine Übereinstimmung ist ab 70% signifikant. Geringere Übereinstimmungen haben keine Bedeutung. Außerdem werden nicht alle Basen in Aminosäuren übersetzt.
14. Beispiel

In der ersten Zeile sind die Identifikationsnummern der gefundenen Nukleinsäure als Hyperlink dargestellt. Ein Klick führt auf die ausführliche Beschreibung, wie sie unter 3. Beispiel weiter oben vorgestellt wurde.
Dahinter erscheint der lateinische Name der biologischen Art, aus dem die Nukleinsäure gewonnen wurde.
Die Anzahl identischer Basen kann man unter Identities ablesen.
Unten werden die zwei verglichenen Basenketten dargestellt. Der obere Strang (Query) entspricht der eingegebenen Sequenz, der untere der gefundenen Sequenz, senkrechte Striche zeigen Identität.
15. Aufgabe
Wieviel Nukleotidstränge mit 200 oder mehr identischen Basen wurden
gefunden?
________
Füllen Sie die folgende Tabelle für die drei Stränge aus, die die größte Übereinstimmung haben:
| Strang |
Artname |
Überein- stimmende Basen/ Gesamtzahl Basen |
|
| Lateinisch | Deutsch | ||
|
|
|||
|
|
|||
|
|
|||
Wie kann man sich die Übereinstimmung der Nukleotidsequenzen verschiedener Porine
a. innerhalb einer Art
b. zwischen Arten erklären?
16. Weitere Aufgaben
Suchen Sie weitere Nukleotidsequenzen. Verwenden Sie keine Umlaute und kein ß. Groß- und Kleinschreibung spielt keine Rolle.
Sie können
- nach Wortanfängen suchen: immunoglob* findet alle Datensätze mit
Wörtern wie immunoglobulin, immunoglobin usw.
- Begriffe mit AND (in Großbuchstaben) verknüpfen: hemoglobin AND homo findet nur Datensätze, die sowohl das Wort hemoglobin als auch das Wort homo aufweisen.
- nach Phrasen suchen: "beta globin" (Anführungszeichen müssen mit eingetippt werden) findet nur Datensätze, die exakt diese Wortkombination enthalten
Suchen Sie folgende Nukleotidsequenzen
- beta-Kette von menschlichem Sichelzellhämoglobin: hemoglobin AND "sickle cell" AND "homo sapiens" AND "beta globin"
- Ionen-Kanalproteine des Menschen: "ion channel" AND "homo sapiens" AND sodium (potassium, calcium)
17. Aminosäuresequenzen
Hier nun eine Aminosäuresequenz aus einem anderen Aquaporin aus Apium graveolens.
Deutscher Name: __________________________________________
YVEPPPAAFIGIDELGKWSFYRALIAEFIATLLFLYITVLTVIGYK
SQSATDPCGGVGILGIAWAFGGMIFVLVYCTAGISGGHINPAVT
Die Aminosäuren werden in folgendem Ein-Buchstaben-Code angegeben:
| A | Alanin | P | Prolin |
| B | Asparaginsäure oder Asparagin | Q | Glutamin |
| C | Cystein | R | Arginin |
| D | Asparaginsäure | S | Serin |
| E | Glutaminsäure | T | Threonin |
| F | Phenylalanin | U | Selenocystein |
| G | Glycin | V | Valin |
| H | Histidin | W | Tryptophan |
| I | Isoleucin | Y | Tyrosin |
| K | Lysin | Z | Glutaminsäure oder Glutamin |
| L | Leucin | X | unbestimmte AS |
| M | Methionin | * | Translationsstop |
| N | Asparagin | - | Lücke unbekannter Länge |
Schreiben Sie die Sequenz der ersten 10 Aminoäsure in ganzen Namen:
Kopieren Sie die Aminosäuresequenz, indem Sie sie mit der Maus markieren und (im Menü des Browsers) mit Bearbeiten/Kopieren in die Zwischenablage befördern.
Gehen Sie dann auf die Startseite der NCBI zurück und klicken Sie auf BLAST.
Auf der aufgerufenen Seite wählen Sie unter Protein BLAST den Menüpunkt Standard protein-protein BLAST [blastp].
Klicken Sie dort in das Feld Search und fügen durch (im Browsermenü) Bearbeiten/Einfügen die Aminosäuresequenz ein.
Standardmäßig wird in den Übersetzungen der GenBank CDS und in den Datenbanken PDB, SwissProt, PIR und PRF gesucht (im Dropdownmenü Choose Database der voreingestellte Eintrag nr.).
Klicken Sie auf den Button Blast.
Auf der folgenden Seite klicken Sie auf Format, um eine formatierte Ausgabe zu erhalten.
Auf der folgenden Seite werden die Datensätze geordnet nach absteigender Übereinstimmung ausgegeben:

Bei diesem Beispiel sieht man, dass mindestens der erste und der vierte Datensatz auch Aquaporine darstellen.
Wenn man auf die blau und unterstrichen dargestellte Zahl am Ende einer Zeile (unter der Überschrift Score (bits), in der Abbildung abgeschnitten) klickt, springt man auf der gleichen Seite hinunter und sieht das Ergebnis des Sequenzvergleichs.
18. Erklärung

Hier wurde der vierte Datensatz ausgesucht, die Aminosäuresequenz stammt aus der Pflanze Samanea saman.
Deutscher Name: _________________________________________
Hinter Identities sieht man, dass 80 von 94 (=85%) der Aminosäuren übereinstimmen.
Darunter steht hinter Query die eingegebene Aminosäuresequenz und hinter Sbjct die gefundene ähnliche Sequenz.
In der Zeile dazwischen wird bei Identität an der Position die Aminosäure noch einmal aufgeführt.
Eine Leerstelle erscheint bei verschiedenen Aminosäuren. Ein + bedeutet
Wie könnten die Nukleotidsequenzen für die ersten 4 Aminosäuren aussehen?
| Code | Name | Mögliche Nukleotidtripletts |
| Y | ||
| V | ||
| E | ||
| P |
Ein Klick auf den blau und unterstrichen dargestellten Anfang der ersten Zeile ruft den vollständigen Datensatz dieser Sequenz auf.
Eine Übereinstimmung ist ab 40% signifikant. Geringere Übereinstimmungen haben keine Bedeutung.
Warum müssen weniger Aminosäuren als Nukleotide für Signifikanz
übereinstimmen?
19. Aufgabe
Hier ist eine Aminosäuresequenz eines weiteren Aquaporins aus
Deutscher Name: __________________________________________
Führen Sie den Sequenzvergleich selber durch.
Füllen Sie die folgende Tabelle für die drei Stränge aus, die die größte Übereinstimmung haben:
| Kette |
Artname |
Überein- stimmende AS/ Gesamtzahl AS |
|
| Lateinisch | Deutsch | ||
| 1 |
|
||
| 2 |
|
||
| 3 |
|
||